snapgene v5.2.0 中文特别版(附安装教程)
SnapGene破解版是一款功能强大的日常分子生物学软件。使用旨在为用户提供完善的计划,可视化和记录您的日常分子生物学程序的解决方案。拥有友好的用户界面,可以方便,全面地记录我们经常生成的所有结构。可在SnapGene中完成所有克隆,并且优化改善你的策略,快速创建质粒图谱,并提供优雅,信息丰富的窗口,用于模拟各种常见的克隆和PCR方法。它节省了大量的时间和金钱,容易操作的可视化和模拟,提前预测可能发生的错误,并提醒用户进行改善,整个过程中,每一个操作都会自动记录,这样的话,用户可以清楚准确的看到你所有的操作内容,并可以通过.dna文件与同事共享丰富的带注释的地图和序列。本次小编带来最新破解版,含破解文件和激活工具,亲测可完美破解激活软件,有需要的朋友不要错过了!
SnapGene是GSL Biotech的产品,GSL Biotech是第一个比笔和纸更易于使用的分子生物学软件。 现在,您实验室中产生的任何DNA都可以记录为电子文件,并与免费软件SnapGene Viewer共享给全世界。
In-Fusion®克隆:一种创建无边界基因连接的通用方法。 SnapGene是第一个模拟此方法的软件。 只需选择要混合的DNA片段,程序就会对其进行设计。
5.0版增加了新功能和显示选项,包括成对比对,从Ensembl数据库导入,对定向TOPO®克隆的支持以及用于与参考DNA序列比对的改进工具。
成对对齐:可以通过局部,全局或半全局比对来分析成对的DNA或蛋白质序列。
从Ensembl导入:现在可以将来自Ensembl基因组浏览器的基因或转录数据直接导入SnapGene。
定向TOPO®克隆:一个新的界面可以模拟定向TOPO®克隆到拓扑异构酶激活的载体中。
灵活对齐参考:与参考DNA序列比对的界面已得到增强。各种显示选项的控件更加直观,并且可以将比对限制在参考序列的指定链或区域内。
可拖动的不对齐端:对于已与参考DNA序列部分比对的序列,现在可以将未比对的末端部分拖出并可视化。
AnzaTM酶:Thermo Fisher(Invitrogen)的AnzaTM酶系统已整合到SnapGene的酶数据库中。
DNA到蛋白质的比对转换:可以翻译DNA比对的选定区域以产生相应的蛋白质比对。
比对cDNA的内含子注释:当cDNA与参考基因组DNA序列比对时,该cDNA可用于创建一个特征,其中的缺口被注释为内含子。
SnapGene 中文破解版安装激活
1,双击SnapGene .exe,安装软件
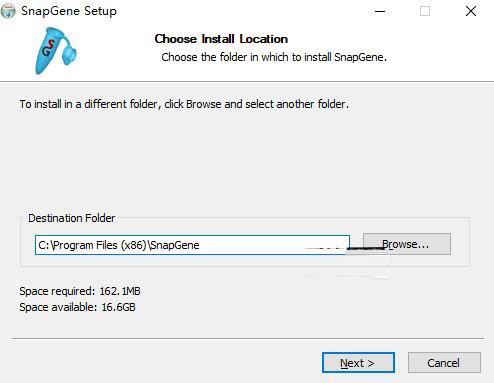
2,软件安装完成后,直接替换SnapGene.exe
默认路径C:\Program Files (x86)\SnapGene
3,安装破解完成,Enjoy
注册信息无需理会,实际是已破解
软件功能
一、想象
1、DNA可视化
查看DNA序列的多个视图。
地图 · 序列 · 酶 · 特征 · 引物 · 历史
自定义酶位点,特征,引物,ORF,DNA颜色等的显示。地图可以是圆形或线性格式。
使用双链模式查看酶位点,具有翻译,引物和DNA颜色的特征。单链模式显示具有彩色特征的紧凑概览。
从一系列酶组中选择。剪切网站可以显示为数字或行,按名称或频率排序。
按名称,位置,大小,颜色,方向性或类型对带注释的要素列表进行排序。复选框在紧凑或完全展开的显示之间切换。
按名称,长度,颜色,结合位点,方向性或解链温度对杂交引物列表进行排序。复选框在紧凑或完全展开的显示之间切换。
查看DNA构建体的自动生成的图形历史记录。祖先序列可以作为单独的文件恢复
2、大序列支持
浏览染色体大小序列。
查看 · 搜索 · 缩放
利用SnapGene的高效数据处理功能,扫描具有数千种注释功能的大型DNA序列。
使用专有的MICA算法立即在染色体内找到序列。
使用多功能控件调整缩放系数和显示区域。
3、蛋白质可视化
查看蛋白质序列的多个视图。
地图 · 序列 · 属性 · 特征 · 历史
自定义区域,站点,键和序列颜色的显示。
使用具有相关特征的1-或3个字母的氨基酸代码查看蛋白质序列。
查看蛋白质的分子量,消光系数,等电点和氨基酸组成。可以对蛋白质的选定部分进行相同的分析。
按名称,位置,大小,颜色或类型对带注释的功能列表进行排序。复选框在紧凑或完全展开的显示之间切换。
查看自动生成的蛋白质序列图形历史记录。祖先蛋白质或DNA序列可以作为单独的文件再生。
4、直观的序列编辑
轻松编辑DNA和蛋白质序列。
标准编辑 · DNA结束
进行插入,删除,替换和大小写更改。复制并粘贴序列时,会自动传输功能。
编辑线性DNA序列的末端以添加或去除突出端或磷酸酯。
5、序列颜色编码
将选定的DNA或氨基酸序列设置为十种颜色之一。
给两条DNA链或蛋白质序列着色。颜色在Map和Sequence视图中都可见。
6、特征注释
自动注释常用功能,或手动注释新功能。
自动特征检测 · 手动特征注释
使用SnapGene广泛的数据库查找DNA序列中的常见特征。您选择的其他功能可以添加到自定义数据库中。
选择DNA或蛋白质序列的一部分,并使用灵活的GenBank兼容控件注释特征。
二、模拟
1、限制性站点指标
确认限制性网站适合克隆。
独特性 · 甲基化敏感性 · 特殊性质
以粗体显示独特的限制性位点,或选择自动定义的Unique Cutters或Unique 6+ Cutters酶组。
不要被愚弄,因为限制性位点被Dam,Dcm或EcoKI甲基化阻断。SnapGene将自动用星号标记该站点。
利用工具提示来避免有问题的限制网站。
2、限制性克隆
可视化克隆过程的所有方面。
如果您已经考虑过程,则模拟只需几秒钟。如果克隆过程存在设计缺陷,则可以捕获并纠正错误。
3、PCR
模拟标准PCR。
使用您自己的引物,或要求SnapGene自动设计引物。产品文件在其历史记录中存储模板和引物。
4、重叠延伸PCR
通过重叠延伸PCR融合片段
最多可组装八个碎片。选择要连接的片段及其方向,SnapGene将设计引物。
5、引物定向诱变
使用诱变引物进行定点诱变。
选择诱变引物,按下按钮查看修饰的质粒。历史颜色突出了突变。
6、网关®克隆
模拟网关® BP克隆或LR克隆,或两者在同一时间。
为方便起见,提供了常见的供体向量和目的向量。选择要组装的片段,SnapGene将设计引物。
7、吉布森大会®
通过融合多达八个片段或通过将多达八个片段插入向量来模拟Gibson Assembly。Gibson Assembly是一种流行的无缝克隆方法。
选择要融合的片段及其方向,SnapGene将设计引物。线性化的载体可以通过酶消化或反向PCR产生。
8、IN-FUSION ®克隆
通过在向量中插入最多八个片段来模拟Clontech的In-Fusion克隆。In-Fusion克隆是一种非常通用的方法,可用于创建无缝基因融合。
选择要融合的片段及其方向,SnapGene将设计引物。线性化的载体可以通过酶消化或反向PCR产生。
9、TA和GC克隆
通过TA或GC克隆捕获PCR产物。
选择传统的TA克隆或高效GC克隆。
为方便起见,提供了常见的TA克隆载体和Lucigen的GC克隆载体。
10、退火Oligos
退火两个寡核苷酸形成双链产物。
使用简单的控件添加突出限制克隆。
三、避免错误
使用SnapGene,确保构造符合您的要求,并确认您获得了所需的序列。
1、阅读基因融合的框架
确保构造中的已转换要素符合框架。
链接翻译 · 警告信息
使用“序列”视图可以快速查看两个已翻译的要素是否在框架中。如果是这样,翻译链接在同一行。如果不是,则翻译在单独的行上。
2、与参考序列对齐
使用强大的对齐工具检查实际构造是否与模拟构造匹配。
地图概述 · 序列详细信息
请参阅对齐序列与参考序列匹配的位置的概述,以及存在差异的位置。
四、自动录制
SnapGene自动记录操作以创建图形历史记录,并将祖先结构存储在最终文件中。
1、全面的“撤销”能力
几乎撤消任何操作。
不要害怕尝试使用SnapGene文件 - 回溯您的步骤很容易。
2、历史和嵌入式祖先
查看构造的图形历史记录。
单击操作以突出显示亲本和产物序列的相关部分,或单击PCR引物名称以查看引物序列。
单击历史记录树中的祖先以重新生成该祖先序列文件,包括其注释和克隆历史记录。
3、历史色彩
使用可选的历史记录颜色来标识序列的最新更改。
详细了解构建体的组装方式,包括结扎的粘性末端。
五、拥有你的数据
SnapGene允许您根据需要阅读和共享文件,同时保持对数据的完全控制。
1、安全文件管理
将SnapGene文件保存在所需的位置。
使用熟悉的安全操作系统来存储和整理SnapGene文件。
2、从其他格式导入
阅读许多常见的文件格式。
不仅可以导入DNA序列,还可以导入注释和注释。我们将继续开发进口商,以确保您不会被锁定为专有文件格式。
3、导出为标准格式
将序列,地图或凝胶图像转换为标准格式,以便与其他软件一起使用。
将序列导出为GenBank或FASTA格式。将地图或模拟琼脂糖凝胶导出为常见的图像格式。
4、使用SnapGene Viewer共享数据
将SnapGene格式的文件发送给可以下载免费SnapGene Viewer的同事或客户。
只需将文件与链接一起发送到SnapGene Viewer即可。收件人将能够看到地图,序列和注释,就像在完整的SnapGene界面中一样。
5、对于开发人员
要将SnapGene整合到您的数据分析或实验室管理软件中,请联系我们以获取有关如何读取和写入SnapGene文件的信息。
六、转换文件格式
开放式信息交换至关重要,因此SnapGene和SnapGene Viewer提供了读取和导出常见文件格式的选项。
从其他格式导入时,目标不仅是捕获DNA序列,还捕获注释和注释
新功能介绍
1、重叠群大会
线性或环状重叠群可以从重叠序列或Sanger序列迹线重新组装。
2、灵活的对齐
现在可以以各种方式导入要对齐的序列。可以提取多个对齐的一部分以生成新的多重对齐,或者可以编辑现有的多个对齐以添加或移除由不同算法生成的对齐。
3、Nicking Enzymes
可以显示Nicking核酸内切酶。当选择跨越从线性序列的末端到切口核酸内切酶位点时,可以通过按Delete删除该单链区域。
4、点特征
现在,DNA序列支持零长度点功能,并在从GenBank,MacVector或Gene Construction Kit导入文件时识别。
5、酶网站突出显示
常用的酶位点可以用黄金突出显示,以便快速识别。
6、协调一致性增强
多重比对的共识序列具有改进的格式,现在可以复制或导出。
7、用于与参考序列对齐的存储编辑
根据流行的需求,当通过调整比对序列的端点编辑与参考DNA序列的比对时,保留和恢复那些编辑。
8、从其他文件格式导入功能
可以将特征导入到BED,GFF3或GTF格式的DNA序列中。
9、从UniProt导入
可以从UniProt数据库导入蛋白质序列记录。
10、进口基因组编译器项目
现在可以在SnapGene中直接打开Genome Compiler项目文件(.gcproj)。对于施工项目文件,在“历史记录”视图中捕获施工历史。
11、引物添加日期
将引物添加到文件时,会记录日期。引物可按添加日期排序。
12、读取和写入对齐文件格式
SnapGene可以将对齐导入SAM / BAM和VectorNTI®.cep格式的参考序列,并可以将对齐导出为SAM / BAM格式的参考序列。
使用帮助
一、调整配置
1、打开SnapGene设置文件
SnapGene设置文件位于:
苹果系统:
〜/库/首选项/ com.gslbiotech.snapgene.plist
视窗:
C:\ Users \用户名\ AppData \漫游\ GSLBiotech \ SnapGene.ini
Linux的:
〜/ .snapgene /的Settings.ini
2、禁用软件更新
要删除所有“检查更新”菜单选项并禁用自动更新,请在“更新”类别中将“禁用”设置为“true”。
3、转移源功能
要确保在克隆时将“源”功能传输到产品,请在“文件”类别中将“transferSourceFeatures”设置为“true”。
二、显示或隐藏酶
1、显示酶
要显示酶,请单击Enzymes → Show Enzymes。
2、隐藏酶
要隐藏酶,请单击酶→隐藏酶。
3、使用侧工具栏切换酶可见性
单击侧工具栏中的“显示酶”按钮也可用于在显示和隐藏所有酶之间切换。
三、选择一套酶套装
Map和Sequence视图中显示的酶可以从预定义或定制酶组列表中选择。
1、选择一套酶套装
要选择酶组,请单击酶→使用酶组→[...]。
2、使用侧工具栏选择酶组
或者,通过单击侧工具栏中的“ 显示酶 ”(限制性位点图标)菜单按钮来选择酶组。
四、手动选择酶
通过名称或编辑现有酶组或使用搜索条件手动选择酶。
1、选择一套酶套装
要选择现有酶组,请单击酶→选择酶,然后在选择:列表中选择酶组名称。
2、按名称选择酶
要在所选酶组中进行搜索,请在搜索框中输入酶名称。
或者,单击酶名称以选择它。
3、添加到酶组
要将选定的酶添加到Chosen Enzymes设置,请单击Add→按钮。
要将原始集中的所有酶添加到Chosen Enzymes集,请单击Add All→按钮。
4、从酶套装中取出
要从Chosen Enzymes设置中删除选定的酶,请单击“ 删除”按钮或按Delete键。
要从Chosen Enzymes设置中删除所有酶,请单击Remove All按钮。
5、按标准选择酶
要根据标准选择酶,请单击“ 选择”复选框。符合所选条件的酶将在“ 选择自:”列表中突出显示
要指定从中选择酶的酶组,请单击“从菜单中选择”按钮。
来指定由其中所选择的酶将减少定义的酶集合,单击酶切割菜单按钮。
要指定剪切站点的数量,请在所选框中键入一个数字。如果之前未指定,则可以在右侧指定剪切站点区域。
要指定酶悬垂类型设置的酶,请单击“ 悬垂”菜单按钮。
要指定识别序列长度,请单击“ 识别序列”菜单按钮。
要仅指定具有回文,不间断或非简并识别序列的酶,请单击相应的框。
下载地址
-
无相关信息